피마 인디언 당뇨병 예측 데이터
- 피마 인디언을 대상으로 당뇨병 발병 여부를 예측
1. 환경 및 데이터 준비
- pandas와 seaborn 라이브러리가 설치되어 있지 않다면 '!pip install 라이브러리'로 먼저 설치
# 필요한 라이브러리 호출
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
- read_csv()
: csv 파일을 불러오는 함수
: pandas에 존재
# 피마 인디언 당뇨병 데이터셋을 불러옴
df = pd.read_csv('./data/pima-indians-diabetes3.csv')
2. 데이터 조사 - 판다스(pandas) 활용
- 판다스(pandas)
: 넘파이의 기능을 포함하면서도 다양한 포맷의 데이터를 다루게 하는 라이브러리
- head()
: 데이터의 처음 n줄을 불러옴
: pandas에 존재
# 처음 5줄을 불러옴
df.head(5)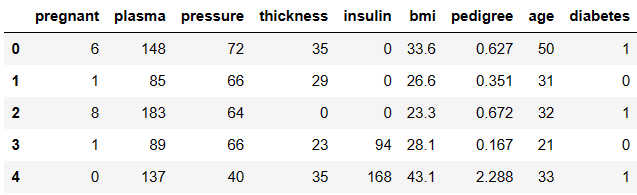
- value_counts()
: 각 컬럼의 값이 몇 개씩 있는지 알려줌
: pandas에 존재
# 정상과 당뇨 환자가 각각 몇 명씩인지 조사
df["diabetes"].value_counts()0 500 # 당뇨에 걸리지 않은 사람
1 268 # 당뇨에 걸린 사람
Name: diabetes, dtype: int64
- describe()
: 정보 별 샘플 수(count), 평균(mean), 표준편차(std), 최솟값(min), 백분위 수로 25%, 50%, 75%, 최댓값(max) 표시
: pandas에 존재
# 각 정보별 특징을 좀 더 자세히 출력
df.describe()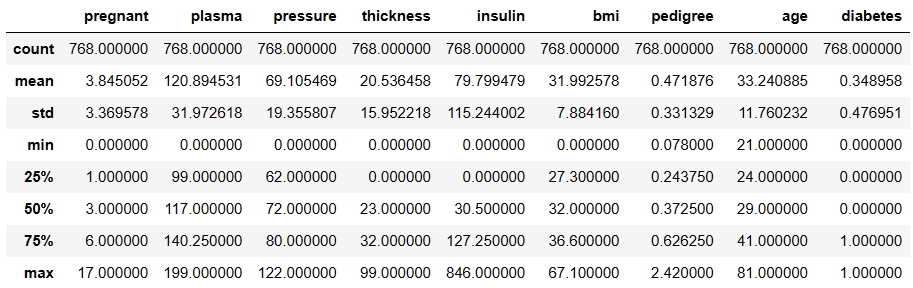
- corr()
: 각 항목이 어느 정도의 상관 관계를 가지고 있는지 출력
: pandas에 존재

3. 데이터 추출 - 시본(seaborn) 활용
- 시본(seaborn) : Matplotlib을 기반으로 구축되었으며, 데이터 시각화 작업을 더 쉽고 정교하게 만들어주는 라이브러리
① 각 항목 간 상관관계를 그래프로 표현하기 위해 색상과 크기 결정
colormap = plt.cm.gist_heat #그래프의 색상 구성을 정함
plt.figure(figsize=(12,12)) #그래프의 크기를 정함
② 각 항목 간 상관관계를 그래프로 나타냄
- heatmap()
: 두 항목씩 짝을 지은 후 각각 어떤 패턴으로 변화하는지 관찰
: 두 항목이 전혀 다른 패턴으로 변화하면 0, 서로 비슷한 패턴으로 변화하면 1에 가까운 값 출력
: seaborn에 존재
- vmap : 색상의 밝기 조절하는 변수
- cmap : 미리 정해진 matplotlib 색상의 설정 값 불러오는 변수
# 그래프의 속성을 결정. vmax의 값을 0.5로 지정해 0.5에 가까울수록 밝은색으로 표시
sns.heatmap(df.corr(),linewidths=0.1,vmax=0.5, cmap=colormap, linecolor='white', annot=True)
plt.show()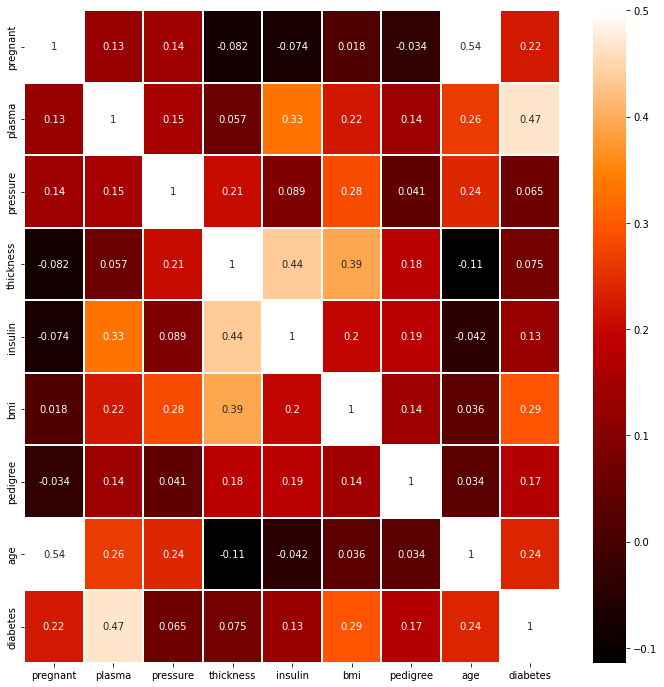
③ plasma 기준으로 diabetes(당뇨) 여부가 어떻게 분포되는지 확인
- hist()
: 히스토그램 그려줌
: matplotlib에 존재
- x축 : 가져오게 될 칼럼 지정
- bins : x축을 몇 개의 막대로 쪼개어 보여줄 것인지 정하는 변수
- barstacked : 여러 데이터가 쌓여있는 형태의 막대바 생성하는 옵션
# plasma를 기준으로 각각 정상과 당뇨가 어느 정도 비율로 분포하는지 살펴봄
plt.hist(x=[df.plasma[df.diabetes==0], df.plasma[df.diabetes==1]], bins=30, histtype='barstacked', label=['normal','diabetes'])
plt.legend()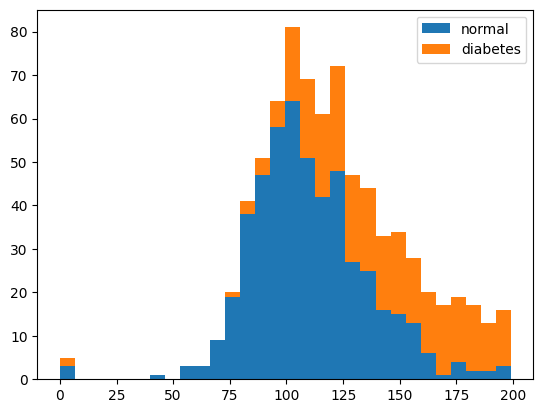
④ BMI 기준으로 diabetes(당뇨) 여부가 어떻게 분포되는지 확인
# BMI를 기준으로 각각 정상과 당뇨가 어느 정도 비율로 분포하는지 살펴봄
plt.hist(x=[df.bmi[df.diabetes==0], df.bmi[df.diabetes==1]], bins=30, histtype='barstacked', label=['normal','diabetes'])
plt.legend()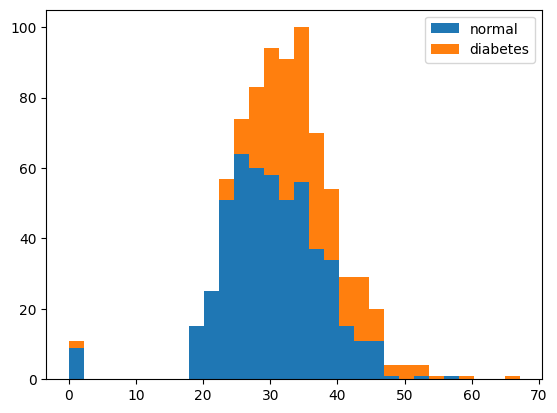
4. 피마 인디언의 당뇨병 예측 실행
- 환경 및 데이터 준비
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
# pandas 라이브러리를 불러옴
import pandas as pd
# 피마 인디언 당뇨병 데이터셋을 불러옴
df = pd.read_csv('./data/pima-indians-diabetes3.csv')- iloc[]
: 데이터 프레임에서 대괄호 안에 정한 범위만큼 가져와 저장
: pandas에 존재
# 세부 정보를 X로 지정
X = df.iloc[:,0:8]
# 당뇨병 여부를 Y로 지정
y = df.iloc[:,8]
- 모델 구조 설정
model = Sequential()
model.add(Dense(12, input_dim=8, activation='relu', name='Dense_1'))
model.add(Dense(8, activation='relu', name='Dense_2'))
model.add(Dense(1, activation='sigmoid',name='Dense_3'))
model.summary()
- Layer : 층의 이름과 유형 나타냄
- Output Shape : 각 층에 몇 개의 출력이 발생하는지 나타냄(샘플의 수, 속성의 수)
- Param : 총 가중치와 바이어스 수의 합(파라미터 수)를 나타냄
- Trainable params : 학습을 진행하면서 업데이트된 파라미터 수
- Non-trainable params : 업데이트 되지 않은 파라미터 수
- 모델 컴파일 및 실행
# 모델 컴파일
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])
# 모델 실행
history=model.fit(X, y, epochs=100, batch_size=5)
'모두의 딥러닝 개정 3판'의 내용과 https://github.com/taehojo/deeplearning의 코드 참고
'AI > 모두의 딥러닝' 카테고리의 다른 글
| [모두의 딥러닝] 16. 과적합(overfitting), 테스트셋 구분, 모델 저장과 재사용 - 초음파 광물 예측 데이터 (0) | 2023.05.26 |
|---|---|
| [모두의 딥러닝] 15. 원 - 핫 인코딩, 소프트맥스(softmax) - 아이리스(iris) 품종 데이터(다중 분류) (0) | 2023.05.25 |
| [모두의 딥러닝] 13. 폐암 수술 환자의 생존율 예측 데이터 분석 (0) | 2023.05.24 |
| [모두의 딥러닝] 12. 기울기 소실(vanishing gradient) 문제, 활성화 함수(ReLU, softplus 등), 고급 경사 하강법(SGD, Adam 등) (0) | 2023.05.22 |
| [모두의 딥러닝] 11. 오차 역전파(back propagation) (2) | 2023.05.22 |

